近日,中国农业科学院作物科学研究所水稻分子设计技术与应用创新团队构建了一个全基因组基因功能单倍型(gcHap)数据集,全面揭示了亚洲栽培稻基因功能单倍型自然变异特征,提出亚洲栽培稻多起源(驯化)假说,并开发了适用于功能单倍型数据全基因组关联分析和全基因组预测的软件包——HAPS。
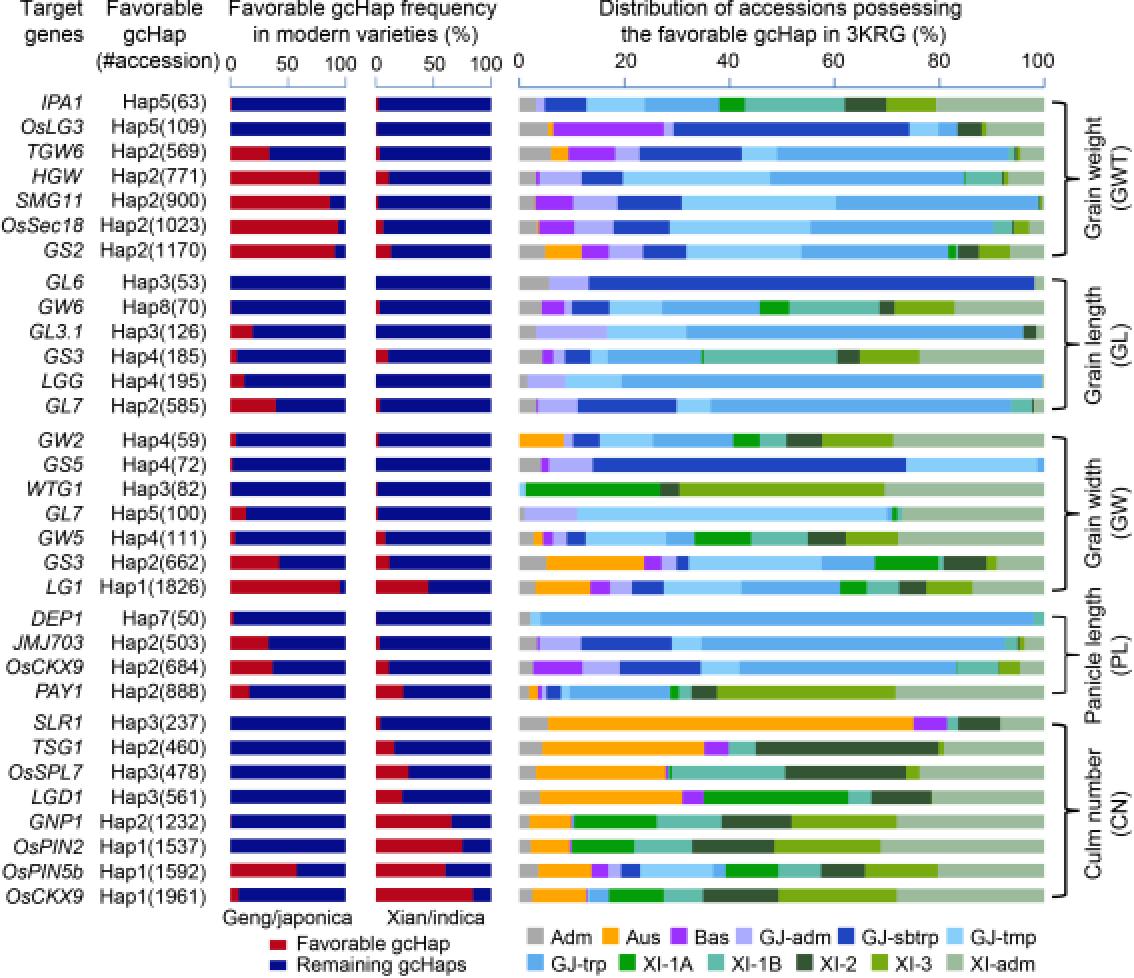
产量相关基因“有利”gcHap在籼粳育成品种中的频率及其在种质资源中的分布(中国农科院供图)
相关研究成果于2月10日在《分子植物(Molecular Plant)》发表。
据介绍,加速育种进程、提升高产潜力和稳产性、兼顾品质改良等,是目前水稻生产的迫切需求。为了加快水稻功能基因组研究与水稻分子育种工作的融合,该团队于2018年牵头完成了“3000份水稻基因组计划”,从三个主要方面揭示了水稻核心种质的基因组多样性,一是超过3200万个高质量SNP;二是3万多个水稻编码基因上的存在-缺失变异;三是超过9.3万个结构变异。
然而,作为水稻种内遗传多样性的另一个重要方面,基因编码区的功能单倍型(功能等位基因)在水稻种质资源中的变异特征尚不清晰。
在该研究中,研究人员根据日本晴参考基因组注释信息,基于3010份水稻核心种质基因编码区的非同义SNP数据,构建了一个由45,963个基因组成的全基因组基因功能单倍型数据集,全面揭示了亚洲栽培稻基因功能单倍型自然变异特征,提出亚洲栽培稻多起源(驯化)假说,评估了现代育种对全基因组功能单倍型多样性的影响,对已克隆基因的有利功能单倍型进行了深入挖掘。
该研究发现,基于功能单倍型数据在全基因组关联分析较SNP数据有更大的功效,在多数性状上具有更高的预测力,并开发了适用于功能单倍型数据全基因组关联分析和全基因组预测的软件包“HAPS”,为今后水稻基因的基础研究和复杂性状有利等位基因发掘提供极大便利,全面助力水稻分子设计育种。同时,也对其他作物群体基因组、功能基因组和设计育种研究具有借鉴意义。













