近日,中国农业科学院农产品加工研究所戴小枫研究员领衔的科研团队发表了首个基于简化基因组测序的棉花黄萎病抗性全基因组关联分析的研究结果,发现了14个抗病新位点,揭示了棉花多基因协同控制黄萎病抗性的分子遗传规律。相关研究成果于3月29日在线发表于植物学国际权威期刊《植物生物技术杂志(Plant Biotechnology Journal)》上。
棉花黄萎病是由大丽轮枝菌(Verticillium dahliae)引起的头号生产病害,也是世界性难题,造成我国棉花年均产量损失15%以上,严重威胁棉花生产安全。生产上一直缺乏抗黄萎病棉花品种,根本原因是对棉花抗黄萎病遗传模式及抗病机制等缺乏系统研究和科学认知,制约了棉花抗黄萎病种质材料的筛选、创制和新品种选育。
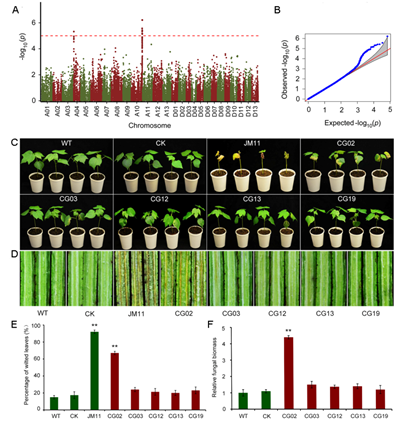
科研团队针对上述棉花抗黄萎病育种的关键问题,系统收集了我国陆地棉优异种质和主栽品种512份,通过全国各地多年多点精准表型鉴定,构建了含299份种质材料的适合于抗病遗传模式解析的自然群体。采用简化基因组测序(SLAF-seq)技术鉴定出覆盖棉花26条染色体的85630个单核苷酸多态性变异位点(SNP),利用全基因组关联分析绘制出棉花全基因组的抗性关联位点图谱,鉴定到17个抗性关联位点、14个抗性新位点,整合基因表达分析和病毒介导的基因沉默(VIGS)技术克隆了一个新的抗黄萎病主效基因。揭示了棉花多基因协同控制黄萎病的抗性分子遗传规律,为后续棉花抗黄萎病功能基因挖掘、分子标记开发和多位点分子标记辅助抗病育种奠定了基础。
该研究得到了中国农科院科技创新工程、国家自然科学基金、公益性行业农业科研专项的资助。加工所博士研究生李廷刚和团队科研骨干马雪峰副研究员为文章的共同第一作者,戴小枫研究员和陈捷胤副研究员为共同通讯作者。(通讯员 杜坷)
原文链接:http://onlinelibrary.wiley.com/doi/10.1111/pbi.12734/abstract

























